分子生物学 知识
开放阅读框
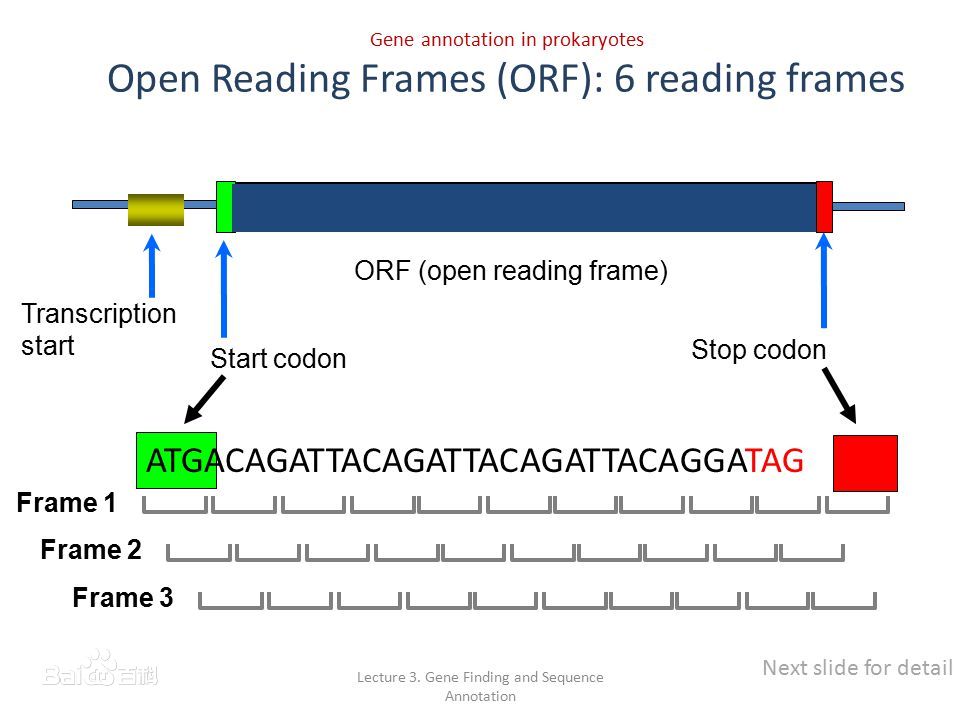
在分子生物学中,开放阅读框(Open Reading Frame, ORF)从起始密码子开始,是DNA序列中具有编码蛋白质潜能的序列,结束于终止密码子连续的碱基序列。
由于密码子(codon)读写起始位点的不同,mRNA序列可能按六种ORF阅读和翻译(每条链三种,对应三种不同的起始位点)。ORF识别则是确定哪种开放阅读框对应真正的多肽编码序列的过程。
在真核生物中,ORF可能跨过外显子,在mRNA中进行拼接。
开放阅读框[open reading frame,ORF] 是结构基因的正常核苷酸序列,从起始密码子到终止密码子的阅读框可编码完整的多肽链,其间不存在使翻译中断的终止密码子。 [1]
在mRNA序列中,每三个连续碱基(即三联“ 密码子”) 编码相应的氨基酸。其中有一个起始密码子AUG和三个终止密码子UAA,UAG,UGA。核糖体从起始密码子开始翻译,沿着mRNA序列合成多肽链并不断延伸,遇到终止密码子时,多肽链的延伸反应终止。由于读写位置不同,ORF在下图链上具有六种可能性。
现在有很多找ORF的软件,包括在线的,如:ORF Finder的功能ORF Finder被用来预测已存在的编码区的小基因序列。它较早应于序列设计,应用优于长片断、高质量的匹配。ORF Finder把提交序列分成六个亚区,并对这六个阅读框分别进行默认,赋予每个亚区一个确定其编码内容的度量, 如果可能将对每一亚区进行进一步分析。每个亚区按照已有的分类结果,被随机提交给查找它们是否编码 蛋白质的特定测试收集器。最后只有那些具有编码潜能的重要区域才被报道。 [1]
- Slightom J L, Durand-Tardif M, Jouanin L, et al. Nucleotide sequence analysis of TL-DNA of Agrobacterium rhizogenes agropine type plasmid. Identification of open reading frames[J]. Journal of Biological Chemistry, 1986, 261(1): 108-121.
高通量测序中,read、contig和Scaffold分别是什么
1.什么是Reads?
高通量测序平台产生的序列就称为reads。
2.什么是Contig?
拼接软件基于reads之间的overlap区,拼接获得的序列称为Contig(重叠群)。
3.什么是Scaffold?
基因组de novo测序,通过reads拼接获得Contigs后,往往还需要构建454 Paired-end库或Illumina Mate-pair库,以获得一定大小片段(如3Kb、6Kb、10Kb、20Kb)两端的序列。基于这些序列,可以确定一些Contig之间的顺序关系,这些先后顺序已知的Contigs组成Scaffold。
Contig N50:Reads拼接后会获得一些不同长度的Contigs.将所有的Contig长度相加,能获得一个Contig总长度.然后将所有的Contigs按照从长到短进行排序,如获得Contig 1,Contig 2,contig 3…………Contig 25.将Contig按照这个顺序依次相加,当相加的长度达到Contig总长度的一半时,最后一个加上的Contig长度即为Contig N50.举例:Contig 1+Contig 2+ Contig 3 +Contig 4=Contig总长度*1/2时,Contig 4的长度即为Contig N50.ContigN50可以作为基因组拼接的结果好坏的一个判断标准.
Scaffold N50:Scaffold N50与Contig N50的定义类似.Contigs拼接组装获得一些不同长度的Scaffolds.将所有的Scaffold长度相加,能获得一个Scaffold总长度.然后将所有的Scaffolds
按照从长到短进行排序,如获得Scaffold 1,Scaffold 2,Scaffold 3…………Scaffold 25.将Scaffold按照这个顺序依次相加,当相加的长度达到Scaffold总长度的一半时,最后一个加上
的Scaffold长度即为Scaffold N50.举例:Scaffold 1+Scaffold 2+ Scaffold3 +Scaffold 4 +Scaffold 5=Scaffold总长度*1/2时,Scaffold 5的长度即为Scaffold N50.Scaffold N50可以作为基因组拼接
的结果好坏的一个判断标准.

