GWAS+WGCNA+基因组组装
全基因组关联研究
全基因组关联研究(Genome-Wide Association Studies,GWAS)是指在全基因组层面上,开展多中心、大样本、反复验证的基因与疾病的关联研究,是通过对大规模的群体DNA样本进行全基因组高密度遗传标记(如SNP或CNV等)分型,从而寻找与复杂疾病相关的遗传因素的研究方法,全面揭示疾病发生、发展与治疗相关的遗传基因。
全基因组关联研究方法的引入,使对遗传流行病的发病预测不再停留在传统的年龄、家族史等“环境性”因素分析,而是通过对人体的全基因组的分析,找出可能导致今后发病的基因,并结合“环境性”因素,得出包括癌症在内的多种流行病的发病率。
以淋巴癌为例,目前国外已通过全基因组遗传流行病研究,找到超过20个可能导致这一病症的基因点位,如果人体的基因组符合这些特征,并结合“种族”这一因素进行综合判断,该个体得淋巴癌的最高可能性将达52%。GWAS为全面系统研究复杂疾病的遗传因素掀开了新的一页,为我们了解人类复杂疾病的发病机制提供了更多的线索。目前,科学家已经在阿尔茨海默、乳腺癌、糖尿病、冠心病、肺癌、前列腺癌、肥胖、胃癌等一系列复杂疾病中进行了GWAS并找到疾病相关的易感基因。
我国科学家也在银屑病、精神病和冠心病等方面开展了GWAS研究并取得成效。如安徽医科大学张学军研究团队就处于GWAS研究的领先水平。
WGCNA-加权基因共表达网络分析
近年来,很多SCI高分文章中都使用了WGCNA分析,那么其分析原理究竟是什么,它可以应用于哪些研究方向,又如何从WGCNA分析结果中挖掘有意义的数据呢?现在就带着这些问题,跟着小诺一起学习探讨吧!
WGCNA概念
WGCNA ,全称为weighted gene co-expression network analysis,即加权基因共表达网络分析。该分析方法旨在寻找协同表达的基因模块(module),并探索基因网络与关注的表型之间的关联关系,以及网络中的核心基因。
WGCNA分析基于两个假设:
相似表达模式的基因可能存在共调控、功能相关或处于同一通路;
基因网络符合无尺度分布
简单举例解释就是样本之间的各个基因是否存在共同表达的模式,例如基因A和基因B是否在某一个阶段中存在相同的表达模式——两者同时上调表达或下调表达。这个方法就是利用这样的思路将样本中基因表达进行分析,探究基因间是否具有共表达的现象,并且根据一定的数值给某一团共表达的基因划分成一个模块,这样聚在一起的不同的团的基因就划分为不同的模块。例如关于调控花青素合成的基因可能就会聚类在同一个模块里面,关于调控叶绿素合成则可能会聚类在另一个模块里面。但是,WGCNA的分析还不止于此,它还可以利用这些模块和表型数据进行聚类,找到这个模块中的核心基因(权重较高的一些基因),也就是hub gene。
WGCNA适用范围
WGCNA一般适合于复杂的转录组数据,推荐5组(或者15个样品)以上的数据。
应用的研究方向主要为:不同器官或组织类型发育调控、同一组织不同发育调控、非生物胁迫不同时间点应答、病原菌侵染后不同时间点应答。
WGCNA术语
在WGCNA分析中有很多相关专业术语,乍看之下可能会让人一头雾水,但其实本质并不复杂,为了后续更好地解读结果数据,就让我们从了解专业术语开始吧!
Co-expression network(共表达网络) :undirected, weighted gene networks,其点代表基因,边代表基因表达相关性,加权(weighted)是指对相关性值进行幂次运算。
Connectivity (连接度):类似于网络中 “度”(degree)的概念,用字母k表示。每个基因的连接度是与其相连的基因的边属性之和。
Module(模块):高度內连的基因集。在无向网络中,模块内是高度相关的基因。
Module eigengene E(模块特征值):模块内所有基因进行主成分分析(PCA),第一主成分的值即为Epigengene。它代表该模块内基因表达的整体水平。
Module membership:给定基因表达谱与给定模型的eigengene的相关性。
Hub gene:关键基因 (连接度最多或连接多个模块的基因)。
TOM (Topological overlapmatrix):把邻接矩阵转换为拓扑重叠矩阵,以降低噪音和假相关,获得的新距离矩阵,这个信息可拿来构建网络或绘制TOM图。
基本原理及方法
WGCNA主要有以下四个步骤:
第一步:构建基因网络;
第二步:分层聚类构建基因模块;
第三步:筛选关键模块
第四步:鉴定关键基因
第一步:构建基因关系网络
基因间相似性(similarity):根据基因在不同样品中的表达情况,用Pearson相关系数计算任意两个基因之间的相关系数(Person Coefficient)。
为了衡量两个基因是否具有相似表达模式,一般需要设置阈值来筛选,高于阈值的则认为是相似的。但是这样如果将阈值设为0.8,那么很难说明0.8和0.79两个是有显著差别的。因此,WGCNA分析时采用相关系数加权值,即对基因相关系数取N次幂,使得网络中的基因之间的连接服从无尺度网络分布(scale-freenetworks),这种算法更具生物学意义。
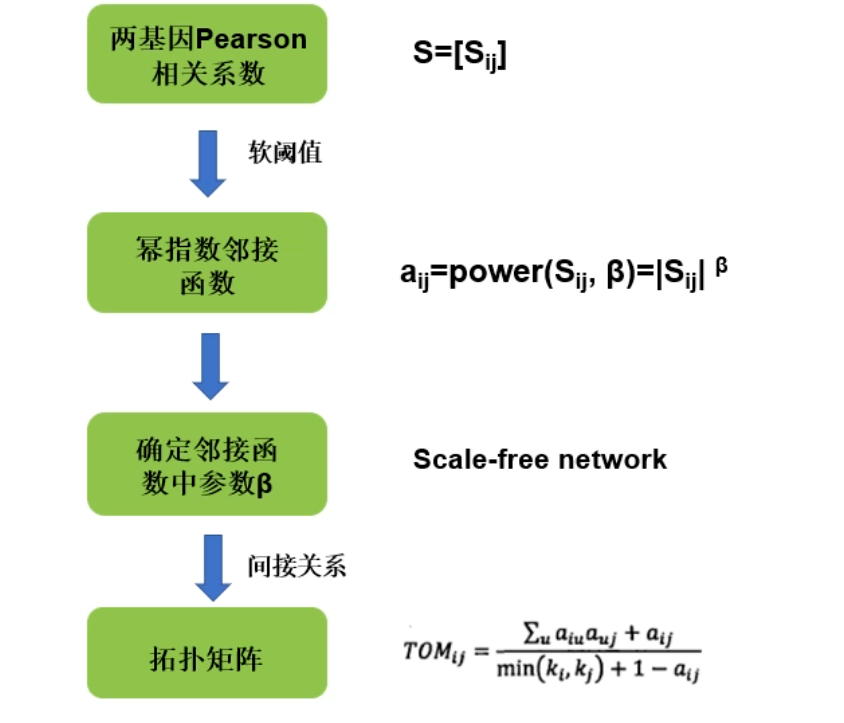
软阈值(soft-thresholding):
β值选取:powers <- c(c(1:10), seq(from = 11, to = 20, by = 1)
sft <- pickSoftThreshold(datExpr, powerVector = powers, verbose = 5)
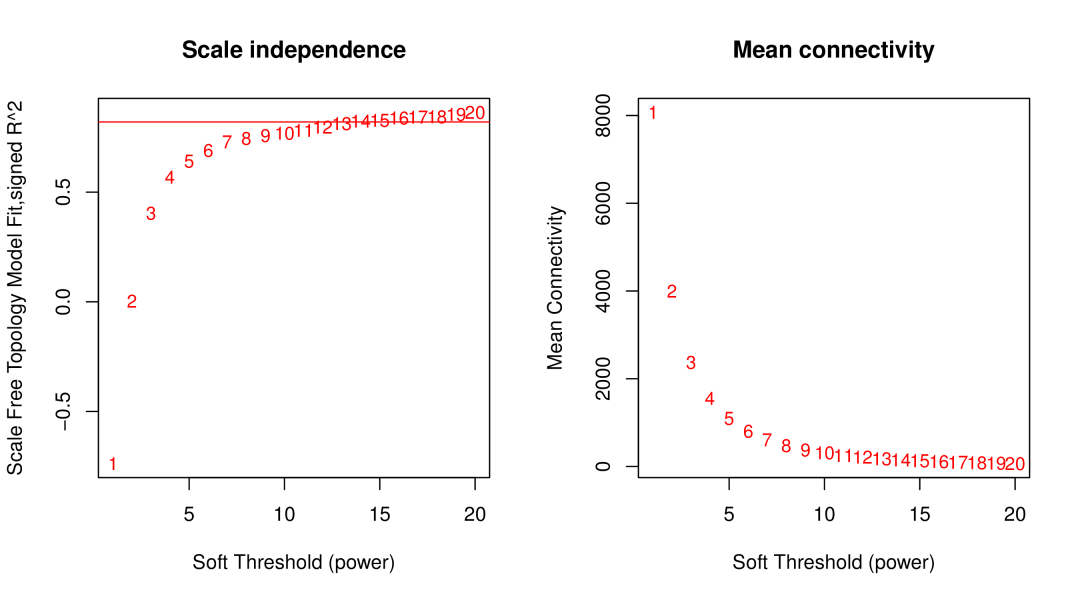
左图横轴均代表权重参数 β, 左图纵轴代表对应的网络中 log(k)与 log(p(k)) 相关系数的平方,红线是此次 WGCNA 分析对应的软阈值和相关系数。相关系数的平方越高,说明该网络越逼近 无网路尺度的分布。
为什么要对2个基因的相关性系数进行幂运算?
基因相关系数经幂函数处理后,少数强相关性不受影响或者影响较小,而相关性弱的取n次幂后,相关性明显下降。如图,对相关性值进行12次幂的运算,数值较小的回趋向0,随着数值大的增大,结果也快速增大。对两个基因的相关系数进行β次幂运算强化了强相关,弱化了弱相关。使基因间的连接网络转化为无尺度网络。前人证明,基因网络接近无尺度网络。
第二步:分层聚类构建基因模块
动态混合剪切法
利用基于TOM值的相异度构建层次聚类建树;建树方法:动态剪切树和静态剪切树。
聚类树的不同分支代表不同的基因模块,不同颜色代表不同的模块。基于基因的加权相关系数,将基因按照表达模式进行分类,将模式相似的基因归为一个模块。这样就可以将几万个基因通过基因表达模式被分成了几十个模块,是一个提取归纳信息的过程。
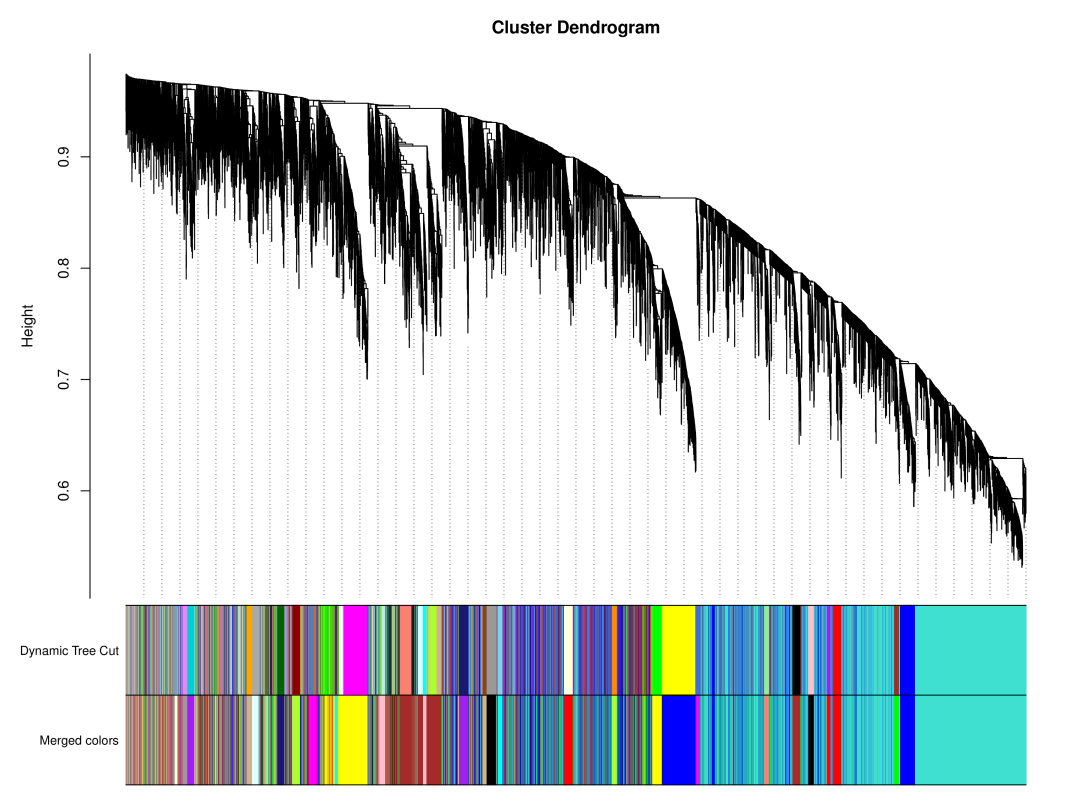 图 模块层次聚类树图
图 模块层次聚类树图
第三步:筛选关键模块
1)根据表达模式分析
我们根据模块特征值(Epigengene,对模块内所有基因进行主成分分析,第一主成分的值即为Epigengene)来计算代表该模块内基因表达的整体水平。如果某模块在样品中特征值的正或负表达较高,说明模块与这个样品关系紧密。
2)模块与样本(表型)相关性
通过计算样本与模块间相关性,我们查看具体那些模块基因与我们关注样本处理紧密相关,从而筛选出重点模块。
3)依据目标基因筛选模块
依据研究目的、前期研究结果和已发表文献,有重点关注的目标基因,可直接筛选目标基因所在的基因模块重点进一步分析。
第四步:鉴定关键基因
TOM值(模块调控系表中的weight值)大于阈值(默认是0.15)的两个基因才认为是相关的,然后计算每个基因的连接度。即先筛选有足够强度的关系,然后计算连接度,连接度越强,说明越处于核心地位;
连接度Connectivity(degree)-连接度:与某个基因连接的所有其他基因的总和,即描述一个基因与其他所有基因的关联程度,一般用K值表示,即我们可以根据K值考前的基因确定hub核心基因。
以上就是WGCNA的分析原理部分,接下来让我们一起结合转录组的WGCNA分析结果来看一下吧。
结果解读
R包分析路径结果中主要有以下文件:
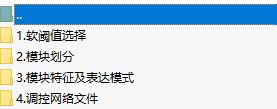
从上面介绍的原理和方法中,可以看出,我们重点关注及挖掘数据主要在“3.模块特征及表达模式”及“4.调控网络文件”。


下面我们重点介绍下主要结果
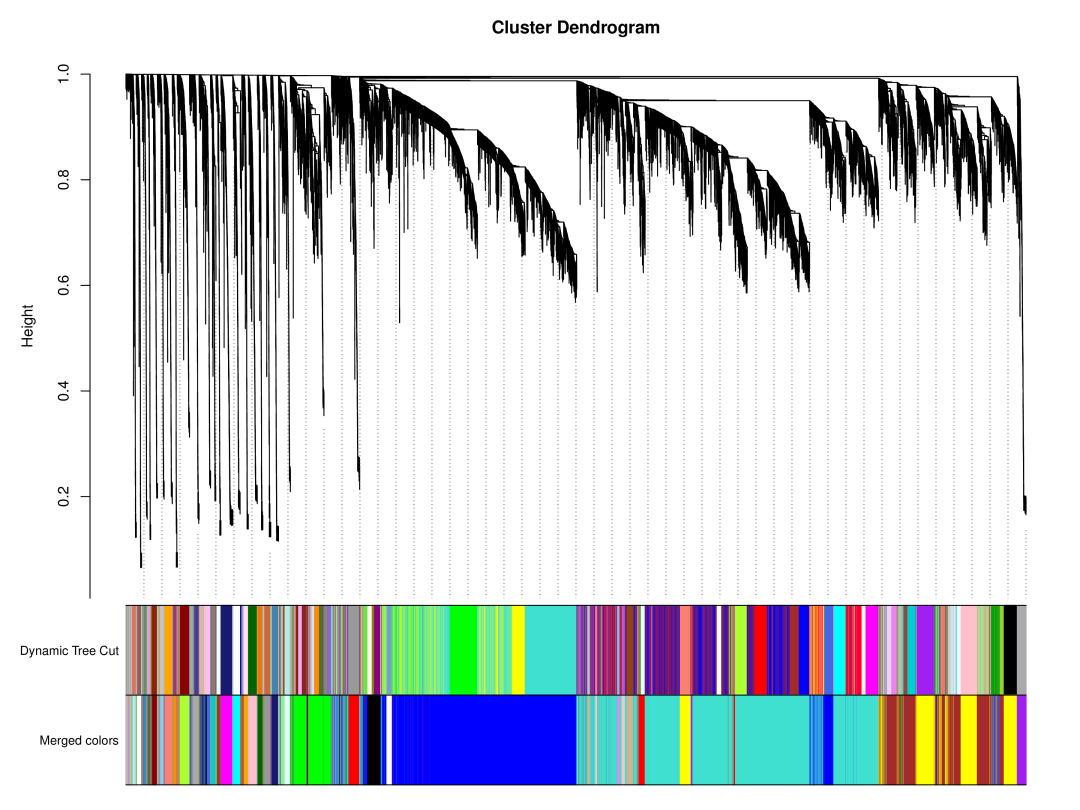
1. 模块层次聚类
WGCNA分析会根据基因间表达量的相关性构建聚类树。图中上半部是网络中的基因聚类树,一个树叶就是一个基因,不同基因模块便是这棵树的树枝。中间部分 Dynamic Tree Cut 是使用动态剪切法获得不同的模块图,其中不同的颜色代表不同的模块。底部 Merged colors 是将相异性系数小于 0.25 的模块合并后的图,其中不同的颜色代表合并后的模块。
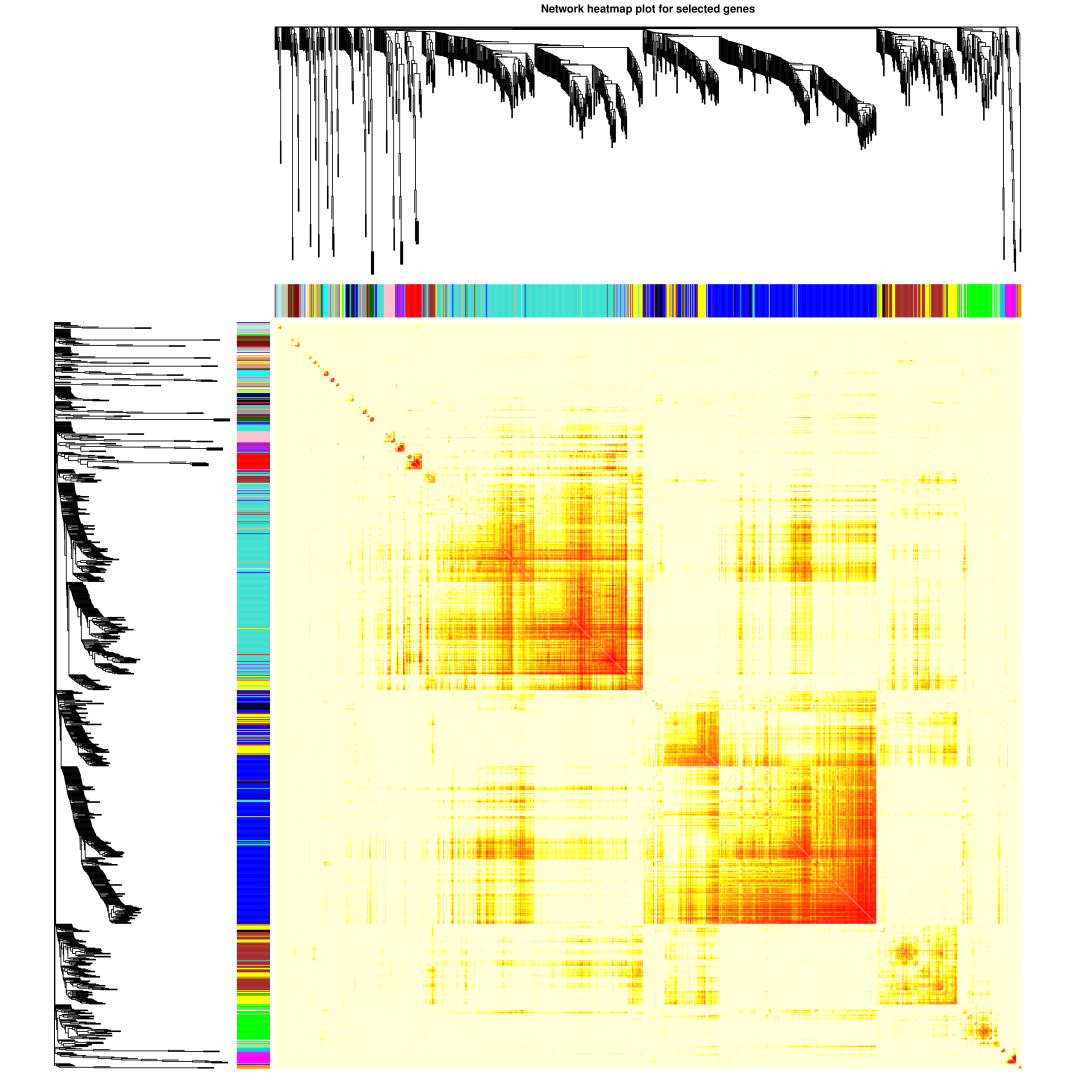
2. 模块基因聚类热图
随机选择 1000 个 基因画拓扑重叠热图,每一树状图代表一个模块,每一个分支代表一个基因,每 个点的颜色越深(白→黄→红)代表行和列对应的两个基因间的连通性越强。
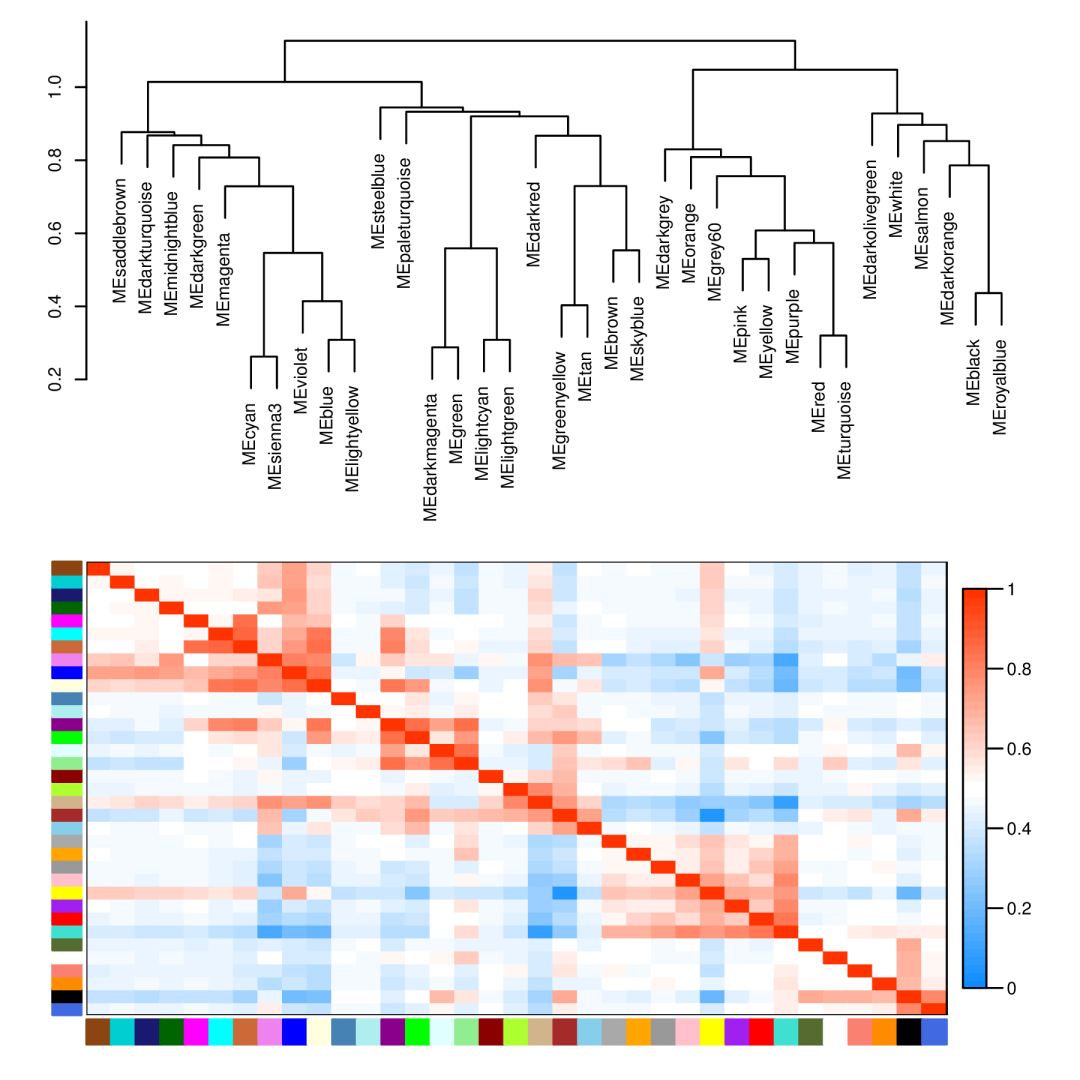
3. 模块与模块之间的相关性热图
该图可分为两部分,上部分是根据模块特征值进行的聚类,通过提取每个模块的特征向量基因作为某一特定模块第一主成分基因,也代表了该模块内基因表达的整体水平,然后对不同模块的特征基因进行了层次聚类,纵坐标的数值是反应的是不同模块间的相似度,越小表示两个模块间相似度越高。下部分是不同模块间的聚类热图,图中每一行和列代表一个模块。方块中颜色越深(越红),相关性越强;方块颜色越浅,相关性越弱。
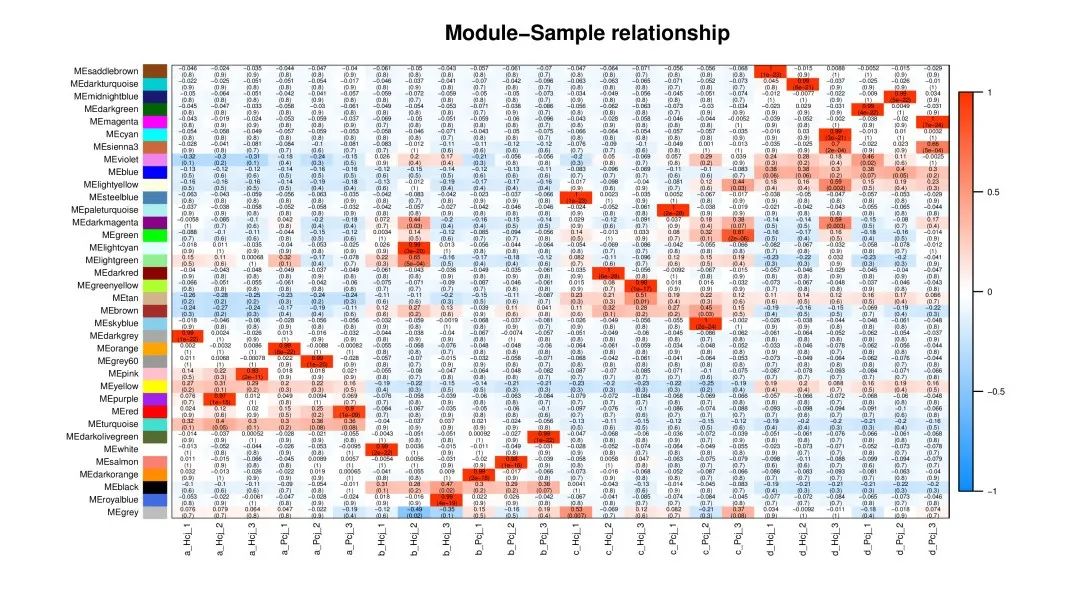
4. 模块与样品的相关性热图
横坐标为样品,纵坐标为模块,每个格子的数字代表模块与样品的相关性,该数值越接近1,表示模块与样品正相关性越强;越接近-1,表示模块与样品负相关性越强。括号里的数字代表显著性 P value,该数值越小,表示显著性越强。
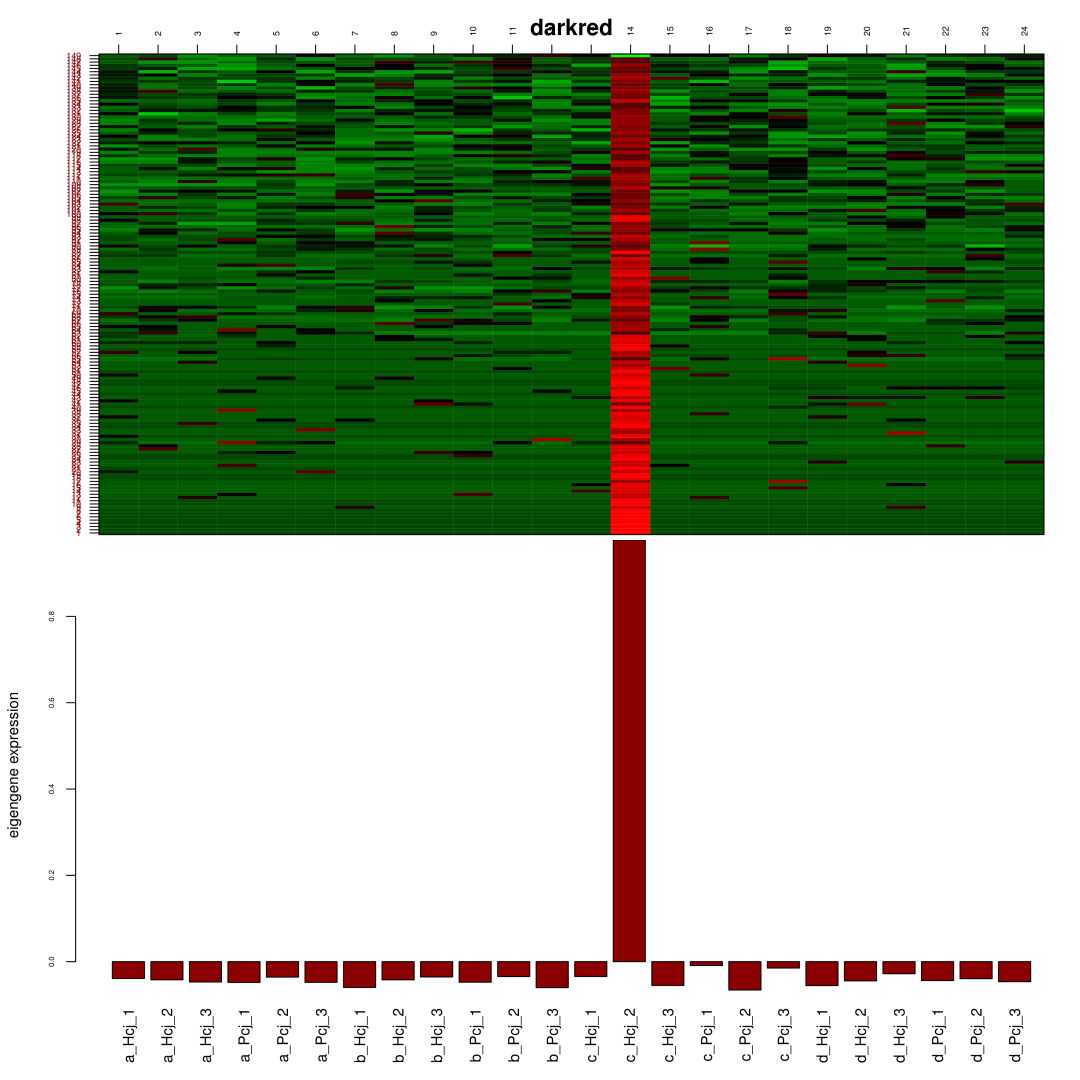
5. 模块内基因表达模式图
该图结果可分为两部分查看,表头注释为模块名,上图左侧为基因名,横坐标为样本名, 上图为模块中基因在不同样本中的表达量热图,红色为高表达,绿色为低表达, 通过上图可直观看出模块中的基因在不同样本中的表达趋势。下图为模块特征值在不同样本中的表达模式,横坐标为样本名,通过下图柱状图的展示,可直观看出哪个样本中的 gene 在该模块下普遍高表达。
6. 各模块基因关系节点文件
模块网络节点关系文件,是每个模块内基因之间相互关联的文件,fromNode是源节点,toNode是靶节点,weight是邻接矩阵的边权重,代表两个节点(基因)之间的连接强度,数值越大代表两个节点(基因)紧密联系或共同表达;direction是连接的方向性,fromAltName和toAltName分别是fromNode和toNode对应的gene_symbol名,需要提供对应的注释文件,否则为NA。
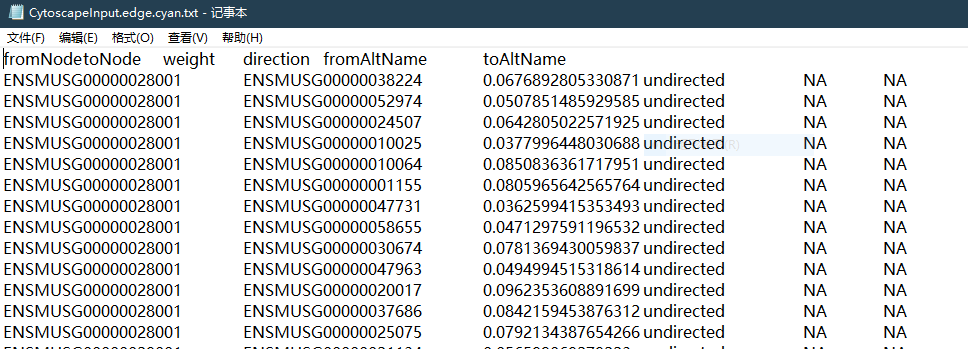
我们可以使用Cytoscape软件利用关系节点文件weight值(TOM值)来绘制网络图。
以上就是WGCNA分析相关内容介绍,想了解更多可参考下方资料:
WGCNA: an R package for weighted correlation network analysis
https://horvath.genetics.ucla.edu/html/CoexpressionNetwork/Rpackages/WGCNA/faq.html
今天的分享就到此结束啦,希望本次的分享可以帮助到大家更好地理解WGCNA分析。如果大家还有其他感兴趣的内容,也可以在评论区留言告诉小诺哦~
基因组组装
前言
基因组组装(Genome assembly)是生物信息学领域的核心问题,基因组组装就是把序列测序产生的读取片段reads经过序列拼接组装,生成基因组的碱基序列。基因组组装软件可根据得到的所有读长组装成基因组。基因组组装这个步骤对于基因组分析是十分关键的,因为目前二代测序技术获得的测序序列一般都较短,需要组装拼接成较长的完整的序列用于进一步分析,例如长序列能提高物种注释分析的准确性。
宏观来说,基因组组装可以分为从头组装(De novo assembly) 和映射比对组装(mapping assembly), 从头组装是指不需要依靠任何已知的基因组信息,反过来,映射比对组装就是需要把测序序列和参考基因组来比对,找到序列的对应位置再进行组装,本文主要讲解的从头组装。 当然两种都有各个的用处,映射比对组装也有一些算法例如BWT算法。
由于目前组装技术的限制和实际情况的复杂性,最终组装得到的序列与真实基因组序列之间仍可能存在差异,甚至只能得到若干条无法进一步连接起来的序列。对组装效果的评价主要依由于据组装序列的连续性、完整性和准确性。连续性要求组装得到的(多条)序列长度尽可能长;完整性要求组装序列的总长度占基因组序列长度的比例尽可能大;准确性要求组装序列与真实序列尽可能符合。
目前基因组组装一般有基于OLC(Overlap-Layout-Consensus, 先重叠后扩展)和基于De Brujin Graph(DBG)两种组装算法,基于OLC的组装方法适合长序列组装,运行依赖的数据结构需要消耗大量的内存,且运行速度比较慢,错误率高,而DBG组装方法内存消耗相对较低,运算速度快,且准确率高。目前主流的基因组装算法都是基于后者改进设计的。
基本概念
在开始之前,有几个名词需要说明下:
- reads:就是我们测序产生的短读序列,通常一代和三代的reads读长在几千到几万bp之间,二代的相对较短,平均是几十到几百bp。
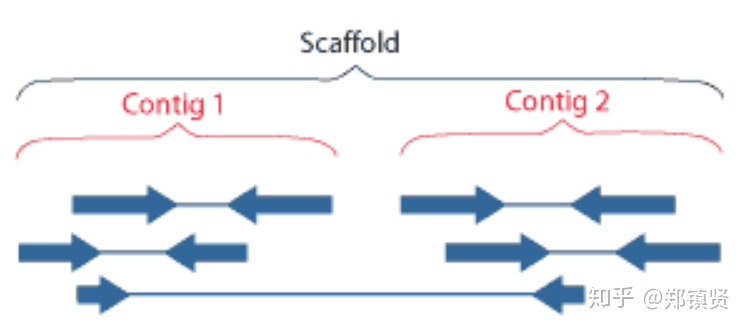
- contig:中文叫做重叠群,就是不同reads之间的overlap交叠区,拼接成的序列就是contig
- scaffold:这是比contig还要长的序列,获得contig之后还需要构建paired-end或者mate-pair库,从而获得一定片段的两端序列,这些序列可以确定contig的顺序关系和位置关系,最后contig按照一定顺序和方向组成scaffold,其中形成scaffold过程中还需要填补contig之间的空缺。
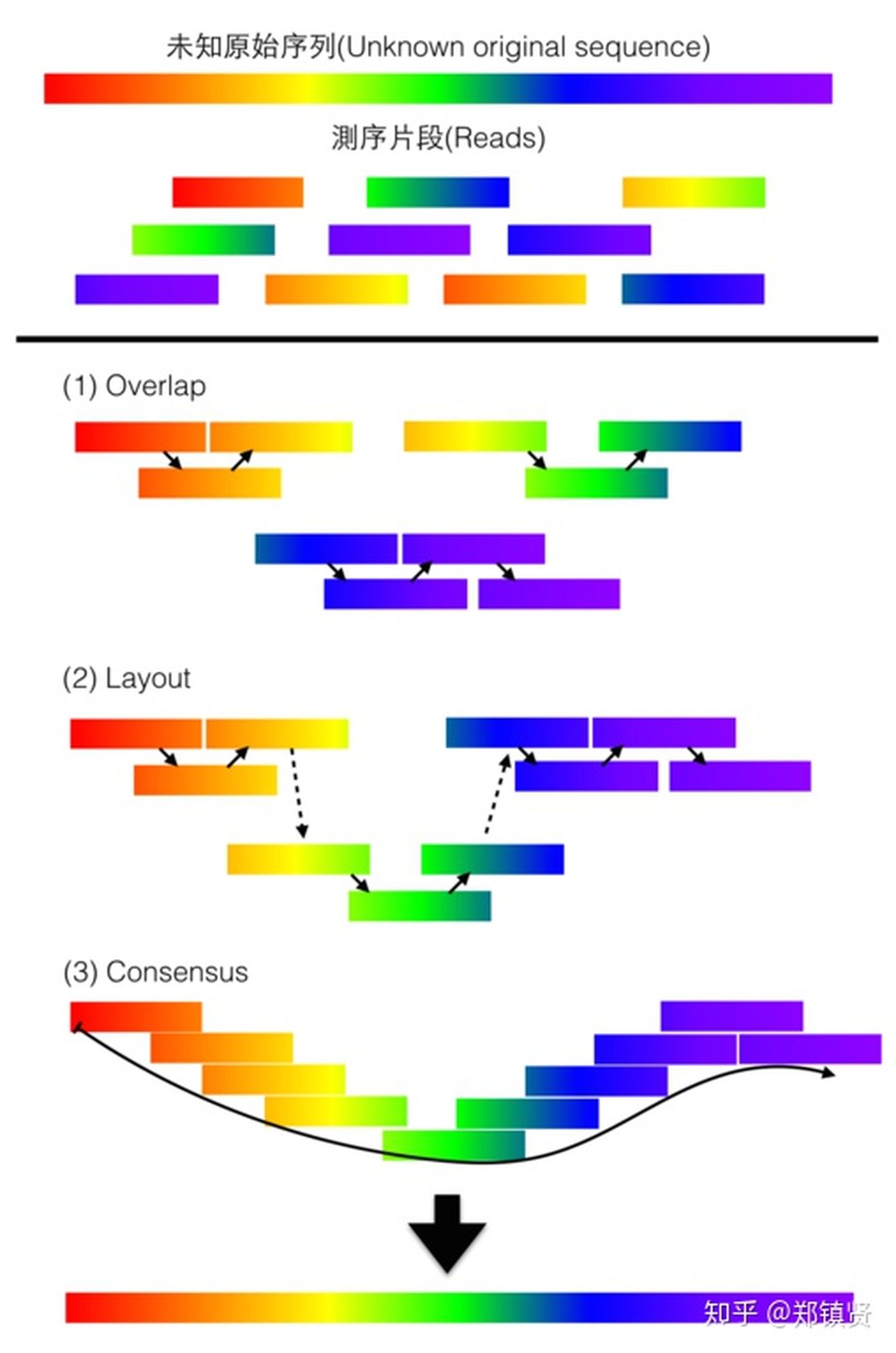
基于OLC算法
OLC组装算法主要这么对一代和三代测序序列因为他们的reads读长相对较长。OLC算法的整体步骤可以分为三步:
- Overlap:对所有reads进行两两比对,找到片段间的重叠信息,一般在比对之前会将reads做下索引,减少计算量。这里需要设定最小重叠长度,如果两个read的最小重叠长度低于一定阈值,那么可以认为两段序列顺序性较差。
- Layout:根据得到的重叠信息将存在的重叠片段建立一种组合关系,形成重叠群,即Contig。Contig进一步排列,生成多个较长的scaffold.
- 根据构成Contig的片段的原始质量数据,在重叠群中寻找一条质量最重的序列路径,并获得与路径对应的序列,即Consensus。通过Consensus的多序列比对算法,就可以获得最终的基因组序列。
基于DBG算法
基于DBG基因组装算法原理图如下:
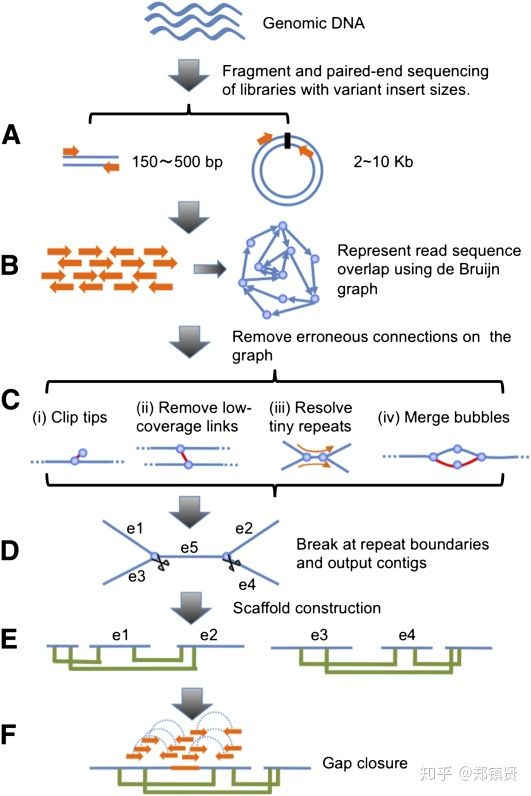
详细的步骤如下:
- 序列k-mer化:对需要测序的片段等大小拆分,即将reads 逐个碱基切分为长度为K的子序列。例如我们的K取3,那么序列ACGTCGA就会被拆分成5个子序列分别是:ACG, CGT, GTC, TCC, CGA。
- de Brujin图构建:de Brujin图是一种有向图,我们将k-mers得到的子序列作为图的节点,如果两个节点有 K-1个共同重叠子集,就把两个节点连接在一起,这样一定程度上已经能够展现出序列的顺序信息了,例如:
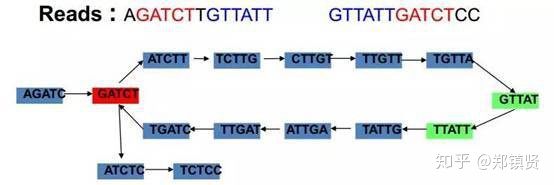
从上面的两个序列得到的de Brujin图可以知道,红色部分为高频的子序列,而绿色的为相对低频的子序列。
- 图结构简化:这里主要简化结构的以下方面:
- 去除低频和低覆盖率的k-mer
- 将小的重复对解开,让每个节点的入度和出度都为1
- 将相似性较高的k-mers合并,也就是bubbles合并成单链
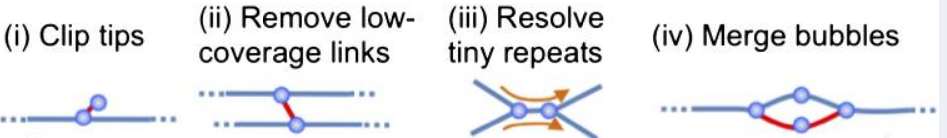
网上有个小哥总结这几个还是不错的
- 测序错误产生的分支:
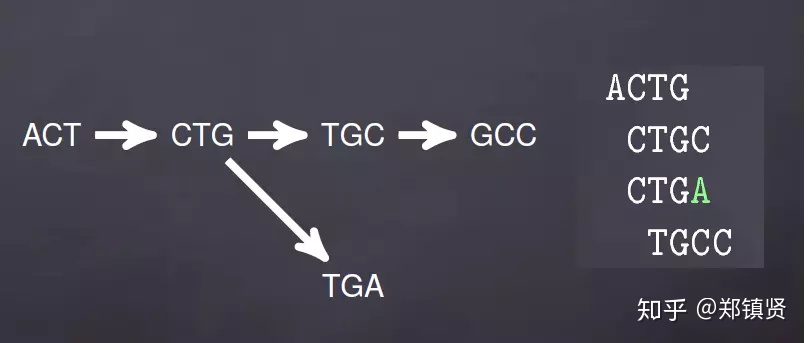
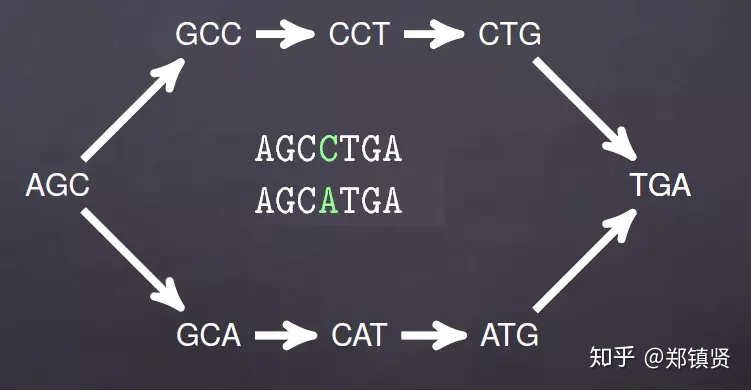
- SNP导致的bubble结构:
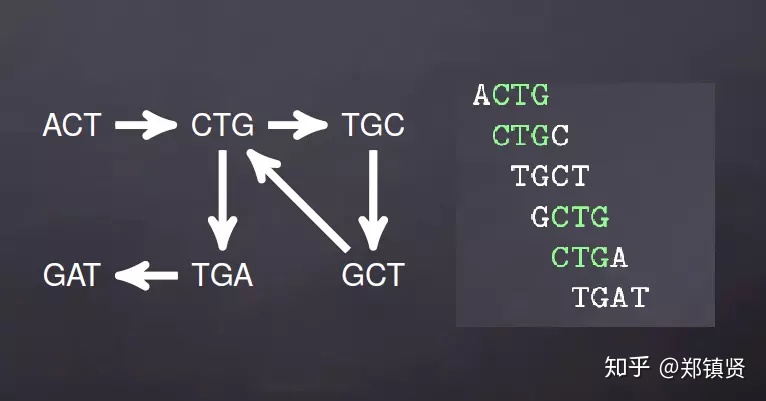
- reads序列重复导致的bubble:
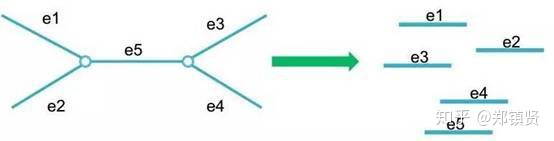
- contigs拆分:通过read的配对末端(pair-end)和环化配对(mate-pair)信息去除一些环结构,最后把一些无法合并的分叉结构再次拆分成多个contigs:
- scaffold构建:这个过程和OLC的比较类似,也是通过contig两端的pair的序列信息,将多个contig连接成scaffold.同时将contig之间的GAP填充,补充方式是通过已经组装的contigs之间的覆盖关系来补充,形成多个没有空缺的scaffold。
- 最终组装:最终是把多个scaffold组装成无GAP的基因组序列
六步教会你基因组组装!
2017-11-14 21:00
序列组装是宏基因组测序分析中的一个重要模块,也是较复杂的部分。不同于一般的基因组组装,其组装出来的是多个微生物基因组序列,这也增加了其复杂度。接下来两期我们将从基因组组装原理和操作方法两个部分为大家全面讲解这部分内容。
基因组组装一般有基于OLC(先重叠后扩展)和基于De Brujin图(DBG)两种组装算法,基于OLC的组装方法适合长序列组装,运行依赖的数据结构需要消耗大量的内存,且运行速度比较慢,错误率高,而DBG组装方法内存消耗相对较低,运算速度快,且准确率高,本期我们主要介绍基于DBG基因组组装算法的基本原理。
基于DBG的基因组组装方法一般分为以下六个步骤:
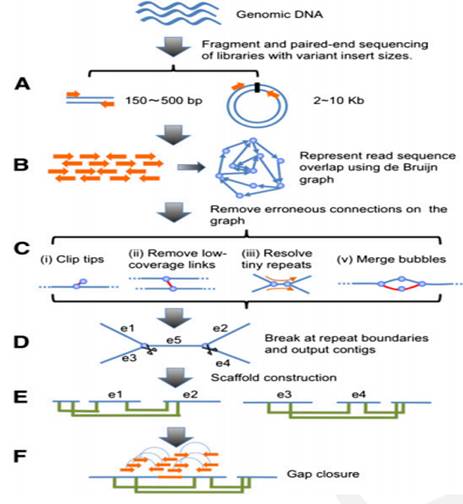
A. 序列k-mer化:对插入片段进行建库测序,下机reads经质控后,对clean reads进行k-mer化,即将reads 逐个碱基开始切分为长度为K的子串;
B. 构建de Brujin图:将上一步得到的所有长度为k的子串即k-mer作为de Brujin图的节点,根据相邻两个K-mer重叠k-1个碱基的原则将该两个顶点(k-mer)有方向的连接起来,构建de Brujin图,如下图所示:
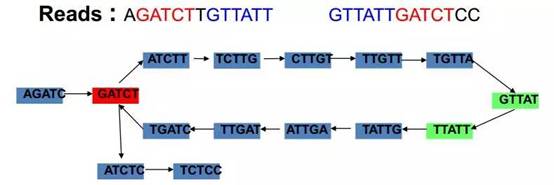
C. DBG简化:去掉无法继续连接和低覆盖度的分支,通常有如下几种情况:
1) 直接删除由于测序错误形成的低频K-mer;

2) 通过短序列将一些很短的重复解开;

3) 如果Kmer1和Kmer2有很高的相似性,将形成的泡状结构合并;

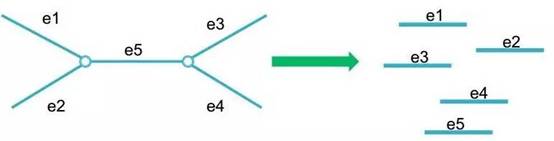
D. 解图获得一致性序列:在简化图的基础上,仍然会因有很多分叉位点无法确定真正的连接关系,因此接下来的每个分叉位点将序列截断,得到contigs;
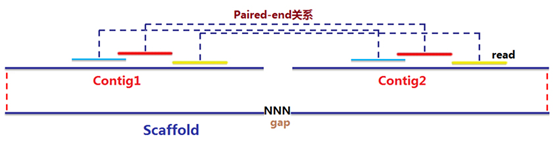
E. 构建scaffold: 将质控后的reads比对回上一步得到的congtigs,利用reads之间的连接关系和插入片段大小信息,将contigs连接成scaffolds;
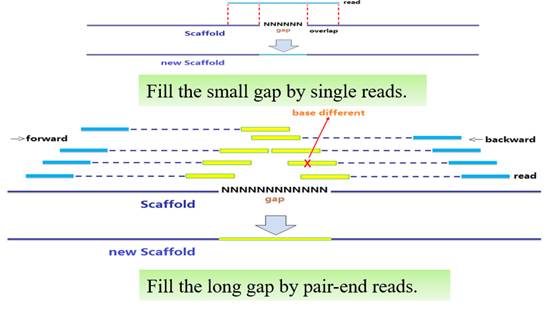
F. Gap Close: 通过PE reads来填补scaffolds内部的Gap,经过Gap填补后,如果还有含N的Gap,则将该条scaffold在Gap处打断,并去掉N,形成最后的scaftigs;
本期讲解的是序列组装的理论部分,大家可以好好消化一下这部分内容,下期将为大家带来具体操作软件及方法,敬请关注!

